Difference between revisions of "Skeletal muscle specific signalling pathways"
(Created page with "==Выделение скоординировано регулируемых групп генов (кластеров) в скелетных мышцах крыс при ат...") |
(→Выделение и анализ регуляторных районов генов, с индуцибельными альтернативными промоторами) |
||
| (24 intermediate revisions by the same user not shown) | |||
| Line 1: | Line 1: | ||
| + | Данные по по изменению генной экспрессии в скелетных мышцах крысы (CAGE) - исследования с вывешиванием крыс за хвост в течение 7 дней и последующим периодом реадаптации (7 дней) к нормальному уровню двигательной активности (моделирование эффектов резкого снижения мышечной активности и последующей реадаптации). Для детальной оценки динамики молекулярных изменений было проведено взятие образцов мышц (''m. soleus'' и ''m. extensor digitorum longus'') через 12 ч, 24 ч, 3 суток и 7 суток после начала мышечной разгрузки и начала реадаптации к нормальному уровню двигательной активности. | ||
| + | |||
==Выделение скоординировано регулируемых групп генов (кластеров) в скелетных мышцах крыс при атрофической разгрузке и реадаптации== | ==Выделение скоординировано регулируемых групп генов (кластеров) в скелетных мышцах крыс при атрофической разгрузке и реадаптации== | ||
| − | Кластеры скоординированно регулируемых генов определялись по экспрессии их промоторов при помощи алгоритма “китайского ресторана” (Qin, 2006), реализованного в платформе BioUML. | + | Кластеры скоординированно регулируемых генов определялись по экспрессии их промоторов при помощи алгоритма “китайского ресторана”(Qin, 2006)<cite>7</cite>, реализованного в платформе BioUML. |
| + | |||
| + | Данные по каждому типу мышц (''m. soleus'' и ''m. extensor digitorum longus'') анализировали независимо. | ||
| + | |||
| + | В результате было выделено по 15 кластеров скоординированно регулируемых генов (табл. [https://docs.google.com/spreadsheets/d/1HVFyQMhkqD4O79QVm6VImcsGKFvs2h04sSMjquNAJ94/edit?usp=sharing 1.3]). Для получения статистически значимых результатов в анализ включали кластеры размером не менее 100 генов. | ||
| + | |||
| + | ==Выделение ключевых молекул: мастер-регуляторов генной экспрессии== | ||
| + | На следующем этапе работы были идентифицированы мастер-регуляторы, регулирующие активацию выявленных сигнальных путей в скелетных мышцах при разгрузке и при реадаптации к нормальному уровню двигательной активности (табл. [https://docs.google.com/spreadsheets/d/1E5ThKF46_xfoObgIda4ZzeG-oAWwKglQgRZwSIsKDoU/edit?usp=sharing 1.4]). Для этого был применен метод поиска сигнальных молекул - мастер-регуляторов (Koschmann et al., 2015)<cite>5</cite>, как было описано выше. | ||
| + | |||
| + | ==Построение возможных путей передачи сигнала в клетке, вовлекающих в себя выявленные транскрипционные факторы и мастер-регуляторы== | ||
| + | На основе выявленных мастер-регуляторов групп скоординированно экспрессированных генов были найдены пути передачи клеточного сигнала, активность которых специфична для клеток мышц, при разгрузке и при реадаптации к нормальному уровню двигательной активности. Для этого к выявленным транскрипционным факторам и мастер-регуляторам был применен метод статистического обогащения биомолекул аннотациями из базы сигнальных путей TRANSPATH. | ||
| + | |||
| + | В число сигнальных путей, активируемых при вывешивании и при восстановлении после него входили: дегенерация мышц с участием γ-секретазы (Rosa de Andrade ''et al'', 2018)<cite>8</cite>, восстановление нормального фенотипа при дистрофии с участием белка Jagged1 (Vieira ''et al'', 2015)<cite>10</cite>, активация синапсов при мышечном сокращении через сигнальный путь BDNF/TrkB (Hurtado ''et al'', 2017)<cite>3</cite> и активация нервной ткани при адаптации скелетной мускулатуры через сигнальный путь NGF-TrkA (Tomlinson ''et al'', 2017) (табл. 1.3)<cite>9</cite> | ||
| + | |||
| + | ==Выделение и анализ регуляторных районов генов, с индуцибельными альтернативными промоторами== | ||
| + | Альтернативные промоторы, индуцибельные в клетках мышечной ткани в условиях реадаптации после вывешивания, были определены в результате кластеризации стартов транскрипции эксперимента CAGE алгоритмом DPI1 (Forrest ''et al'', 2014)<cite>2</cite>, с использованием робастных порогов (на старт транскрипции приходится не менее 11 прочтений и его TPM-нормированный скор не менее 1), и выделения уникальных промоторов в сравнении с промоторами генома крысы, определенными в ходе проекта FANTOM5. | ||
| + | |||
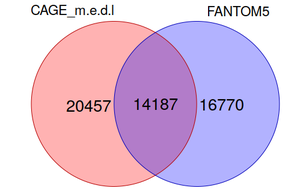
| + | Всего в результате анализа данных эксперимента CAGE по реадаптации крыс после вывешивания было найдено 34644 промоторов в мышце длинного разгибателя пальцев (''m. extensor digitorum longus''), 14187 из которых пересекались с промоторами генома крысы из проекта FANTOM5 (найденными в различных тканях крысы в базальном состоянии), а 20457 - были уникальными для клеток ''m. extensor digitorum longus'' (обнаружены в нашем эксперименте) (рис. 1.2). | ||
| + | |||
| + | [[File:Figure1.2.png|300px|Figure 1.2]] | ||
| + | |||
| + | Рис. 1.2. Сопоставление промоторов, обнаруженных в нашем исследовании в m. extensor digitorum longus крысы (показано красным), с промоторами, аннотированных для генома крысы в проекте FANTOM5 (суммарные данные по разным тканям, показано синим). | ||
| + | |||
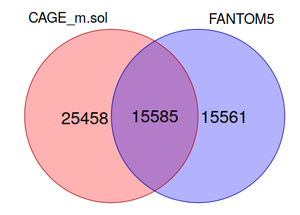
| + | Также было найдено 40632 промотора, в камбаловидной мышцы (<i>m. soleus</i>), 15585 из которых пересекались с промоторами генома крысы из проекта FANTOM5 (найденными в различных тканях крысы в базальном состоянии), а 25458 - были уникальными для клеток m. soleus (обнаружены в нашем эксперименте) (рис. 1.3) | ||
| + | |||
| + | [[File:Figure1.3.png|300px|Figure 1.3]] | ||
| + | |||
| + | <b>Рис. 1.3.</b> Сопоставление промоторов, обнаруженных в нашем исследовании в ''m. soleus'' крысы (показано красным), с промоторами, аннотированных для генома крысы в проекте FANTOM5 (суммарные данные по разным тканям, показано синим). | ||
| + | |||
| + | В промоторах активных при реадаптации в мышцах ''m. extensor digitorum longus'' и ''m. soleus'' были найдены сайты связывания транскрипционных факторов, с использованием базы данных TRANSFAC, статистически значимо обогащенные по сравнению с сайтами, найденными в промоторах, представленные в базе FANTOM5. | ||
| + | |||
| + | В силу недостаточной аннотации генома крысы (по сравнению с геном человека), найденные мотивы сайтов были аннотированы ортологичными генами человека (табл. 1.5 и 1.6) | ||
| + | |||
| + | Функциональная аннотация найденных факторов транскрипции проводилась с помощью анализа обогащения по базе данных TRANSPATH (табл. 1.7 и 1.8). | ||
| + | |||
| + | В результате анализа обогащения новых индуцибельных промоторов в ''m. extensor digitorum longus'' крысы, активированных при реадаптации после вывешивания, были выявлены процессы ассоциированные с сигнальным путем аполипопротеина E (apoE) (Kang et al, 2008)<cite>4</cite>, активированным оксистеролом, а также с транспортом липидов, активируемым печеночным рецептором X (LXR ) (Archer et al, 2014)<cite>1</cite>. | ||
| + | |||
| + | В результате анализа обогащения новых индуцибельных промоторов в ''m. soleus'' крысы, активированных при реадаптации после вывешивания, были выявлены сигнальные пути ядерного фактора активированных T-клеток NFAT5, управляющие миграцией и дифференцировкой миобластов при регенерации скелетных мышц (O'Connor et al, 2007)<cite>6</cite>. | ||
| + | |||
| + | Всего в мышцах ''m. extensor digitorum longus'' и ''m. soleus'' при реадаптации было активно 46923 промотора, большинство которых - 29510 - были активны в обеих мышцах. Интересно, что количество промоторов, активных только в камбаловидной мышце (11880) более чем в два раза превышало количество промоторов, активных только в разгибателе пальцев (5533) (рис. 1.4) | ||
| + | |||
| + | [[File:Skeletal human - Figure1.4.png|300px|Skeletal human - Figure 1.4]] | ||
| + | |||
| + | Рис. 1.4. Сопоставление промоторов, обнаруженных в нашем исследовании в m. soleus крысы (показано красным), с промоторами обнаруженными в нашем исследовании в m.extensor digitorum longus (показано синим). | ||
| + | |||
| + | ==Литература== | ||
| + | <biblio> | ||
| + | #1 Archer A, Laurencikiene J, Ahmed O, Steffensen KR, Parini P, Gustafsson JÅ, Korach-André M. Skeletal muscle as a target of LXR agonist after long-term treatment: focus on lipid homeostasis. Am J Physiol Endocrinol Metab. 2014. V. 306(5). P. E494-502. | ||
| + | |||
| + | #2 Forrest A.R., Kawaji H., Rehli M., Baillie J.K., De Hoon M.J., Haberle V., Lassmann T., Kulakovskiy I.V., Lizio M., Itoh M., Andersson R. A promoter-level mammalian expression atlas. Nature. 2014 . V. 507. № 7493. P. 462. | ||
| + | |||
| + | #3 Hurtado E, Cilleros V, Nadal L, Simó A, Obis T, Garcia N, Santafé MM, Tomàs M, Halievski K, Jordan CL, Lanuza MA, Tomàs J. Muscle Contraction Regulates BDNF/TrkB Signaling to Modulate Synaptic Function through Presynaptic cPKCα and cPKCβI. Front Mol Neurosci. 2017. V. 10. P. 147. | ||
| + | |||
| + | #4 Kang J, Albadawi H, Patel VI, Abbruzzese TA, Yoo JH, Austen WG Jr, Watkins MT. Apolipoprotein E-/- mice have delayed skeletal muscle healing after hind limb ischemia-reperfusion. J Vasc Surg. 2008. V. 48(3). P. 701-8. | ||
| + | |||
| + | #5 Koschmann J., Bhar A., Stegmaier P., Kel A., Wingender E. “Upstream analysis”: an integrated promoter-pathway analysis approach to causal interpretation of microarray data. Microarrays. 2015. V. 4. № 2. P. 270-286. | ||
| + | |||
| + | #6 O'Connor RS, Mills ST, Jones KA, Ho SN, Pavlath GK. A combinatorial role for NFAT5 in both myoblast migration and differentiation during skeletal muscle myogenesis. J Cell Sci. 2007 Jan 1;120(Pt 1):149-59. doi: 10.1242/jcs.03307. Epub 2006 Dec 12. PMID: 17164296. | ||
| + | |||
| + | #7 Qin Z.S. Clustering microarray gene expression data using weighted Chinese restaurant process. Bioinformatics. 2006. V. 22. № 16. P. 1988-1997. | ||
| + | |||
| + | #8 Rosa de Andrade, I., Corrêa, S., Fontenele, M., de Oliveira Teixeira, J. D., Abdelhay, E., Costa, M. L., Mermelstein, C., γ-Secretase Inhibition Induces Muscle Hypertrophy in a Notch-Independent Mechanism. Proteomics. 2018. V. 18(3-4). doi: 10.1002/pmic.201700423. | ||
| − | + | #9 Tomlinson RE, Li Z, Li Z, Minichiello L, Riddle RC, Venkatesan A, Clemens TL. NGF-TrkA signaling in sensory nerves is required for skeletal adaptation to mechanical loads in mice. Proc Natl Acad Sci U S A. 2017. V. 114(18). P. E3632-E3641. | |
| − | + | #10 Vieira NM, Elvers I, Alexander MS, Moreira YB, Eran A, Gomes JP, Marshall JL, Karlsson EK, Verjovski-Almeida S, Lindblad-Toh K, Kunkel LM, Zatz M. Jagged 1 Rescues the Duchenne Muscular Dystrophy Phenotype. Cell. 2015. V. 163(5). P. 1204-1213. | |
| + | </biblio> | ||
Latest revision as of 21:04, 12 March 2021
Данные по по изменению генной экспрессии в скелетных мышцах крысы (CAGE) - исследования с вывешиванием крыс за хвост в течение 7 дней и последующим периодом реадаптации (7 дней) к нормальному уровню двигательной активности (моделирование эффектов резкого снижения мышечной активности и последующей реадаптации). Для детальной оценки динамики молекулярных изменений было проведено взятие образцов мышц (m. soleus и m. extensor digitorum longus) через 12 ч, 24 ч, 3 суток и 7 суток после начала мышечной разгрузки и начала реадаптации к нормальному уровню двигательной активности.
Contents
- 1 Выделение скоординировано регулируемых групп генов (кластеров) в скелетных мышцах крыс при атрофической разгрузке и реадаптации
- 2 Выделение ключевых молекул: мастер-регуляторов генной экспрессии
- 3 Построение возможных путей передачи сигнала в клетке, вовлекающих в себя выявленные транскрипционные факторы и мастер-регуляторы
- 4 Выделение и анализ регуляторных районов генов, с индуцибельными альтернативными промоторами
- 5 Литература
Выделение скоординировано регулируемых групп генов (кластеров) в скелетных мышцах крыс при атрофической разгрузке и реадаптации
Кластеры скоординированно регулируемых генов определялись по экспрессии их промоторов при помощи алгоритма “китайского ресторана”(Qin, 2006)[1], реализованного в платформе BioUML.
Данные по каждому типу мышц (m. soleus и m. extensor digitorum longus) анализировали независимо.
В результате было выделено по 15 кластеров скоординированно регулируемых генов (табл. 1.3). Для получения статистически значимых результатов в анализ включали кластеры размером не менее 100 генов.
Выделение ключевых молекул: мастер-регуляторов генной экспрессии
На следующем этапе работы были идентифицированы мастер-регуляторы, регулирующие активацию выявленных сигнальных путей в скелетных мышцах при разгрузке и при реадаптации к нормальному уровню двигательной активности (табл. 1.4). Для этого был применен метод поиска сигнальных молекул - мастер-регуляторов (Koschmann et al., 2015)[2], как было описано выше.
Построение возможных путей передачи сигнала в клетке, вовлекающих в себя выявленные транскрипционные факторы и мастер-регуляторы
На основе выявленных мастер-регуляторов групп скоординированно экспрессированных генов были найдены пути передачи клеточного сигнала, активность которых специфична для клеток мышц, при разгрузке и при реадаптации к нормальному уровню двигательной активности. Для этого к выявленным транскрипционным факторам и мастер-регуляторам был применен метод статистического обогащения биомолекул аннотациями из базы сигнальных путей TRANSPATH.
В число сигнальных путей, активируемых при вывешивании и при восстановлении после него входили: дегенерация мышц с участием γ-секретазы (Rosa de Andrade et al, 2018)[3], восстановление нормального фенотипа при дистрофии с участием белка Jagged1 (Vieira et al, 2015)[4], активация синапсов при мышечном сокращении через сигнальный путь BDNF/TrkB (Hurtado et al, 2017)[5] и активация нервной ткани при адаптации скелетной мускулатуры через сигнальный путь NGF-TrkA (Tomlinson et al, 2017) (табл. 1.3)[6]
Выделение и анализ регуляторных районов генов, с индуцибельными альтернативными промоторами
Альтернативные промоторы, индуцибельные в клетках мышечной ткани в условиях реадаптации после вывешивания, были определены в результате кластеризации стартов транскрипции эксперимента CAGE алгоритмом DPI1 (Forrest et al, 2014)[7], с использованием робастных порогов (на старт транскрипции приходится не менее 11 прочтений и его TPM-нормированный скор не менее 1), и выделения уникальных промоторов в сравнении с промоторами генома крысы, определенными в ходе проекта FANTOM5.
Всего в результате анализа данных эксперимента CAGE по реадаптации крыс после вывешивания было найдено 34644 промоторов в мышце длинного разгибателя пальцев (m. extensor digitorum longus), 14187 из которых пересекались с промоторами генома крысы из проекта FANTOM5 (найденными в различных тканях крысы в базальном состоянии), а 20457 - были уникальными для клеток m. extensor digitorum longus (обнаружены в нашем эксперименте) (рис. 1.2).
Рис. 1.2. Сопоставление промоторов, обнаруженных в нашем исследовании в m. extensor digitorum longus крысы (показано красным), с промоторами, аннотированных для генома крысы в проекте FANTOM5 (суммарные данные по разным тканям, показано синим).
Также было найдено 40632 промотора, в камбаловидной мышцы (m. soleus), 15585 из которых пересекались с промоторами генома крысы из проекта FANTOM5 (найденными в различных тканях крысы в базальном состоянии), а 25458 - были уникальными для клеток m. soleus (обнаружены в нашем эксперименте) (рис. 1.3)
Рис. 1.3. Сопоставление промоторов, обнаруженных в нашем исследовании в m. soleus крысы (показано красным), с промоторами, аннотированных для генома крысы в проекте FANTOM5 (суммарные данные по разным тканям, показано синим).
В промоторах активных при реадаптации в мышцах m. extensor digitorum longus и m. soleus были найдены сайты связывания транскрипционных факторов, с использованием базы данных TRANSFAC, статистически значимо обогащенные по сравнению с сайтами, найденными в промоторах, представленные в базе FANTOM5.
В силу недостаточной аннотации генома крысы (по сравнению с геном человека), найденные мотивы сайтов были аннотированы ортологичными генами человека (табл. 1.5 и 1.6)
Функциональная аннотация найденных факторов транскрипции проводилась с помощью анализа обогащения по базе данных TRANSPATH (табл. 1.7 и 1.8).
В результате анализа обогащения новых индуцибельных промоторов в m. extensor digitorum longus крысы, активированных при реадаптации после вывешивания, были выявлены процессы ассоциированные с сигнальным путем аполипопротеина E (apoE) (Kang et al, 2008)[8], активированным оксистеролом, а также с транспортом липидов, активируемым печеночным рецептором X (LXR ) (Archer et al, 2014)[9].
В результате анализа обогащения новых индуцибельных промоторов в m. soleus крысы, активированных при реадаптации после вывешивания, были выявлены сигнальные пути ядерного фактора активированных T-клеток NFAT5, управляющие миграцией и дифференцировкой миобластов при регенерации скелетных мышц (O'Connor et al, 2007)[10].
Всего в мышцах m. extensor digitorum longus и m. soleus при реадаптации было активно 46923 промотора, большинство которых - 29510 - были активны в обеих мышцах. Интересно, что количество промоторов, активных только в камбаловидной мышце (11880) более чем в два раза превышало количество промоторов, активных только в разгибателе пальцев (5533) (рис. 1.4)
Рис. 1.4. Сопоставление промоторов, обнаруженных в нашем исследовании в m. soleus крысы (показано красным), с промоторами обнаруженными в нашем исследовании в m.extensor digitorum longus (показано синим).
Литература
- Qin Z.S. Clustering microarray gene expression data using weighted Chinese restaurant process. Bioinformatics. 2006. V. 22. № 16. P. 1988-1997.
- Koschmann J., Bhar A., Stegmaier P., Kel A., Wingender E. “Upstream analysis”: an integrated promoter-pathway analysis approach to causal interpretation of microarray data. Microarrays. 2015. V. 4. № 2. P. 270-286.
- Rosa de Andrade, I., Corrêa, S., Fontenele, M., de Oliveira Teixeira, J. D., Abdelhay, E., Costa, M. L., Mermelstein, C., γ-Secretase Inhibition Induces Muscle Hypertrophy in a Notch-Independent Mechanism. Proteomics. 2018. V. 18(3-4). doi: 10.1002/pmic.201700423.
- Vieira NM, Elvers I, Alexander MS, Moreira YB, Eran A, Gomes JP, Marshall JL, Karlsson EK, Verjovski-Almeida S, Lindblad-Toh K, Kunkel LM, Zatz M. Jagged 1 Rescues the Duchenne Muscular Dystrophy Phenotype. Cell. 2015. V. 163(5). P. 1204-1213.
- Hurtado E, Cilleros V, Nadal L, Simó A, Obis T, Garcia N, Santafé MM, Tomàs M, Halievski K, Jordan CL, Lanuza MA, Tomàs J. Muscle Contraction Regulates BDNF/TrkB Signaling to Modulate Synaptic Function through Presynaptic cPKCα and cPKCβI. Front Mol Neurosci. 2017. V. 10. P. 147.
- Tomlinson RE, Li Z, Li Z, Minichiello L, Riddle RC, Venkatesan A, Clemens TL. NGF-TrkA signaling in sensory nerves is required for skeletal adaptation to mechanical loads in mice. Proc Natl Acad Sci U S A. 2017. V. 114(18). P. E3632-E3641.
- Forrest A.R., Kawaji H., Rehli M., Baillie J.K., De Hoon M.J., Haberle V., Lassmann T., Kulakovskiy I.V., Lizio M., Itoh M., Andersson R. A promoter-level mammalian expression atlas. Nature. 2014 . V. 507. № 7493. P. 462.
- Kang J, Albadawi H, Patel VI, Abbruzzese TA, Yoo JH, Austen WG Jr, Watkins MT. Apolipoprotein E-/- mice have delayed skeletal muscle healing after hind limb ischemia-reperfusion. J Vasc Surg. 2008. V. 48(3). P. 701-8.
- Archer A, Laurencikiene J, Ahmed O, Steffensen KR, Parini P, Gustafsson JÅ, Korach-André M. Skeletal muscle as a target of LXR agonist after long-term treatment: focus on lipid homeostasis. Am J Physiol Endocrinol Metab. 2014. V. 306(5). P. E494-502.
- O'Connor RS, Mills ST, Jones KA, Ho SN, Pavlath GK. A combinatorial role for NFAT5 in both myoblast migration and differentiation during skeletal muscle myogenesis. J Cell Sci. 2007 Jan 1;120(Pt 1):149-59. doi: 10.1242/jcs.03307. Epub 2006 Dec 12. PMID: 17164296.